深圳大学汪业军联合北京大学刘伟团队解析光滑念珠菌临床分离株基因组进化特征和耐药新机制
近日,北京大学第一医院刘伟团队、深圳大学医学部汪业军团队以“Genome analysis and machine learning-based feature selection strategy reveal potential drug-resistance determinants in Nakaseomyces glabratus”为题在Emerging Microbes & Infections发表论文,深入解析了侵袭性念珠菌病的重要病原体——光滑念珠菌的基因组及其进化特征,并且鉴定了新的关键耐药驱动基因突变。
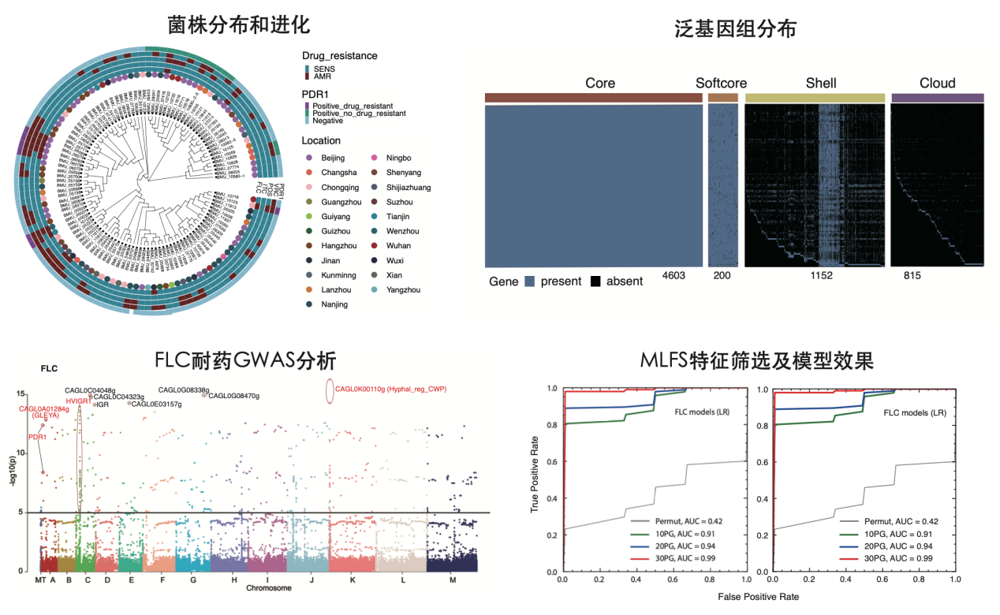
研究从2002-2021年间的中国20多个城市病人的血液和腹水标本总分离了109株光滑念珠菌,其中将近一半(48.6%)对三唑类抗真菌药物不敏感,8株(7.3%)对卡泊芬净不敏感。对这些光滑念珠菌进行全基因组测序和泛基因组学分析,鉴定出6770个泛基因组家族,其中29%为附属基因组且缺乏功能注释,表明光滑念珠菌基因组的高度可塑性。光滑念珠菌的基因组存在广泛的基因扩增,扩增的基因经常与光滑念珠菌参考菌株的基因同源性低,而与其它真菌或非真菌基因表现出高度同源,提示光滑念珠菌和其它物种(甚至细菌)间存在物种间基因水平转移(HGT)。
通过基因组关联分析(GWAS),鉴定出光滑念珠菌唑类耐药性相关的基因组标签。为了更加精准地确定光滑念珠菌的耐药性,团队开发了一种基于机器学习的特征筛选策略(MLFS),利用该策略筛选出唑类耐药菌株核心基因组耐药决定子,其中包括已证实的可以导致光滑念珠菌对唑类药物耐药的基因譬如PDR1的突变及一些未知基因的变化。对于新鉴定的唑类耐药标记,团队进一步利用自行建立的基于CRISPR-Cas9系统的光滑念珠菌基因操作方法展开实验验证及机制分析。
本研究可以加深对光滑念珠菌基因组及其可塑特性的理解,开发的基于机器学习的特征筛选策略(MLFS)可用于病原真菌的耐药性预测,并为确定光滑念珠菌耐药性决定子提供新的靶点和线索。本项目由国家重点研发计划资助支持。
论文全文链接:https://doi.org/10.1080/22221751.2025.2595789


用户登录
还没有账号?
立即注册