医学部朱卫国教授团队在Cell Reports上发表DNA损伤修复过程中R-Loop被精准识别并清除的研究成果
2024年2月,医学部卡尔森国际肿瘤中心朱卫国特聘教授团队在《Cell Reports》杂志上发表题为“The ARID1A-METTL3-m6A axis ensures effective RNase H1-mediated resolution of R-loops and genome stability”的研究论文,揭示了R-loop中RNA发生的m6A甲基化修饰对于RNase H识别R-loop并驱动R-Loop清除起到十分关键的作用。医学部助理教授张俊,研究生陈凤为本研究共同第一作者,朱卫国特聘教授为通讯作者。

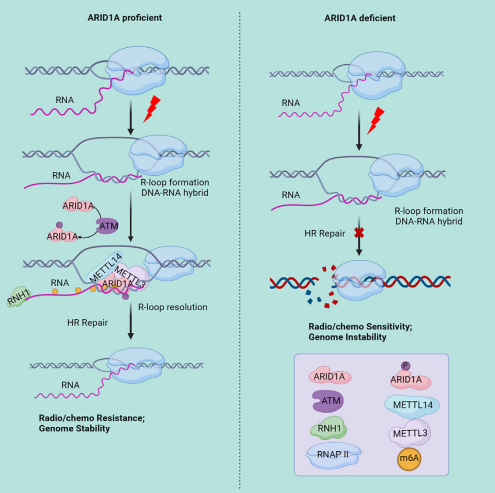
R-Loop是新转录生成的RNA链与DNA模板链形成的三链结构,这种结构在生物学中具有重要的功能,但R-Loop在特定基因组位点的过度累积会引发基因组的不稳定性。细胞内存在多种机制防范R-Loop的过度积累,其中RNase H被公认为降解R-Loop的关键分子。然而,目前人们尚不清楚R-Loop是如何被RNase H精确地识别和分解,并最终达到维持基因组稳定性的目的。
本研究首先证实了RNase H1更倾向结合带有m6A甲基化修饰的R-Loop,并推动R-Loop的分解以促进 DSB 的 DNA 末端切除,从而确保基因组的稳定性。机制上,该团队鉴定出ARID1A在响应DNA损伤应激时快速识别R-Loop并进一步招募RNA甲基转移酶METTL3/14,从而驱动R-Loop RNA 发生m6A修饰。功能研究显示ARID1A及METTL3的缺失会导致R-Loop积累,同时ARID1A-METTL3信号轴缺陷的细胞对基因毒性疗法更加敏感。鉴于异常表达的ARID1A与肿瘤患者对基因毒性疗法的耐药性有关,这些发现为探索针对ARID1A异常表达癌症的治疗提供了潜在新策略 (具体工作模式图如下)。
本研究得到国家自然科学基金,广东省自然科学基金以及深圳市医学研究基金等项目的资助。
原文链接:https://www.sciencedirect.com/science/article/pii/S2211124724001074。










用户登录
还没有账号?
立即注册